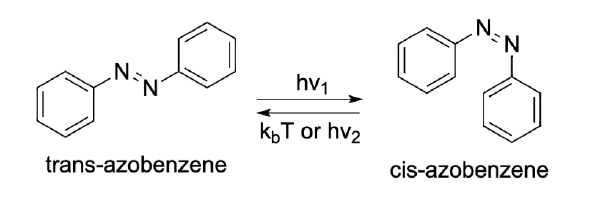
Amber Umbrella Sampling(伞状采样)教程
Umbrella Sampling 伞状采样
参考链接:AMBER高级教程A17: 伞形采样计算平均力势PMF MoBioChem
前言
通常我们可能想要知道自由能随特定反应坐标的分布情况. 这种分布被称为平均力势, 它对识别过渡态, 中间产物和终点的相对稳定性是非常有用的, 一开始人们可能认为能够通过运行一次MD模拟来产生自由能随特定反应坐标的变化, 然后查看样品采样的概率. 然而, 通常感兴趣的能量位垒是kbT的数倍, 因此MD模拟将仍然处在它一开始的局部最小值或者跨越不同的最小值, 但极少会采样到过渡态. 采样的缺乏使得我们不能产生一个精确的PMF. 事实上, 甚至对于很小的系统我们也需要超过一毫秒的MD去获得可用的采样. 这显然很难达到, 因此需要使用更好的方法.本文所需文件
具体步骤
系统准备
偶氮苯结构文件获取ZINC数据库 下载偶氮苯(Azobenzene)的 mol2文件
在本机创建工作路径并处理偶氮苯
1 2 3 4 5 6 7 antechamber -i Azobenzene.mol2 -fi mol2 -o lig.com -fo gcrt antechamber -i lig.com -fi gcrt -o lig.mol2 -fo mol2 -c bcc -at gaff parmchk2 -i lig.mol2 -f mol2 -o lig.frcmod
! 使用两次antechamber是为了确保文件内格式的正确,以顺利的模拟。
antechamber命令解释:
-i -o: 输入输出文件-fi -fo: 指定输入输出文件格式-c bcc: 指定电荷类型为AM1-BCC-at gaff: 指定原子类型
初始MD
在进行伞状采样之前,我们需要对偶氮苯系统进行平衡,使偶氮苯处于平衡状态之后,在进行伞状采样。
1 2 3 4 5 cp ../1_init/lig.mol2 . cp ../1_init/lig.frcmod . tleap -f tleap.in
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 source leaprc.gaff2source leaprc.water.tip3pMOL=loadmol2 lig.mol2 loadamberparams lig.frcmod saveoff MOL lig.lib solvateoct MOL WAT 10 saveamberparm MOL system.parm7 system.rst7 savepdb MOL system.pdb quit
上述步骤,我们将偶氮苯放在了一个边长为10A的八面体盒子中,并对其溶剂化。生成的下一步使用的文件为system.parm7和system.rst7
1 vmd system.parm7 system.rst7
Minimization
1 sander -O -i min.in -p system.parm7 -c system.rst7 -o min.out -r min.rst7 -inf min.info
1 2 3 4 5 6 7 8 Minimization &cntrl imin=1 maxcyc=4000 ncyc=1000 ntpr=2 /
Heating
1 sander -O -i heat.in -p system.parm7 -c min.rst7 -o heat.out -x heat.crd -r heat.rst7 -inf heat.info
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 Heat &cntrl imin=0, ntx=1, irest=0, nstlim=10000, dt=0.002, ntf=2, ntc=2, tempi=0.0, temp0=300.0, ntpr=1, ntwx=10, ioutfm=0, iwrap=1, cut=8.0, ntb=1, ntp=0, ntt=3, gamma_ln=2.0, nmropt=1, ig=-1, / &wt type ='TEMP0' , istep1=0,istep2=8000, value1=0.0,value2=300.0, / &wt type ='TEMP0' , istep1=8000, istep2=10000, value1=300.0, value2=300.0, / &wt type ='END' /
Production
1 2 3 sander -O -i prod.in -p system.parm7 -c heat.rst7 -o prod.out -r prod.rst7 -x prod.crd -inf prod.info & process_mdout.perl prod.out
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 Prod &cntrl imin=0 irest=0 ntx=7 ntb=2 ntp=1 barostat=1 pres0=1.0 taup=2.0 cut=8.0 ntc=2 ntf=2 tempi=300.0 ntt=3 gamma_ln=1.0 nstlim=40000 dt=0.002 ntpr=200 ntwx=200 ntwr=200 ntxo=1 ioutfm=0 iwrap=1 /
Umbrella Sampling
限制二面角的MD
1 sander -O -i prod.in -p system.parm7 -c prod.rst7 -r prod_180.rst7 -x prod_180.crd -inf prod_180.info &
1 2 3 4 5 6 7 8 Harmonic restraints for 180 deg &rst iat=4,5,6,7, r1=60.0,r2=180.0,r3=180.0,r4=300.0, rk2=200,rk3=200, /
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 Prod &cntrl imin=0 irest=0 ntx=7 ntb=2 ntp=1 barostat=1 pres0=1.0 taup=2.0 cut=8.0 ntc=2 ntf=2 tempi=300.0 ntt=3 gamma_ln=1.0 nstlim=2000 dt=0.002 ntpr=10 ntwx=10 ntwr=20 ntxo=1 ioutfm=0 nmropt=1 / &wt type ='DUMPFREQ' , istep1=50, &end &wt type ='END' , &end DISANG=disang DUMPAVE=dihedral_180.dat
伞状采样
进行采样需要多个窗口,创建脚本,生成伞状采样的窗口。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 bash umbrella.sh for i in { 1..9 }do mv md$i .crd md0$i .crd done parm system.parm7 trajin *.crd 1 last 20 autoimage :MOL trajout final.crd go exit vmd system.parm7 final.crd xmgrace dihedral{1..180}.out
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 delta=$(echo "-3" | bc) rini=$(echo "180" | bc) limit =61n=1 while [ $n -le $limit ]do r=$(echo "$rini +$delta *($n -1)" | bc) rmin=$(echo "$r -30.0" | bc) rmax=$(echo "$r +30.0" | bc) [ -e md$r .in ] && rm md$r .in [ -e dihedral$r .dat ] && rm dihedral$r .dat echo umbrellatest >> md$r .in echo \&cntrl >> md$r .in echo imin=0 >> md$r .in echo irest=1 >> md$r .in echo ntx=7 >> md$r .in echo ntb=2 >> md$r .in echo ntp=1 >> md$r .in echo barostat=1 >> md$r .in echo pres0=1.0 >> md$r .in echo taup=2.0 >> md$r .in echo cut=8.0 >> md$r .in echo ntc=2 >> md$r .in echo ntf=2 >> md$r .in echo tempi=300.0 >> md$r .in echo temp0=300.0 >> md$r .in echo ntt=3 >> md$r .in echo gamma_ln=1.0 >> md$r .in echo nstlim=4000 >> md$r .in echo dt=0.002 >> md$r .in echo ntpr=10 >> md$r .in echo ntwx=5 >> md$r .in echo ntwr=5 >> md$r .in echo ntxo=1 >> md$r .in echo ioutfm=0 >> md$r .in echo nmropt=1 >> md$r .in echo / >> md$r .in echo \&wt >> md$r .in echo type =\'DUMPFREQ\' , >> md$r .in echo istep1=1, >> md$r .in echo \&end >> md$r .in echo \&wt >> md$r .in echo type =\'END\' , >> md$r .in echo \&end >> md$r .in echo DISANG=dihedral$r .dat >> md$r .in echo DUMPAVE=dihedral$r .out >> md$r .in echo Harmonic restraints for $r dihedral >> dihedral$r .datecho \&rst >> dihedral$r .datecho iat=4,5,6,7 >> dihedral$r .datecho r1=$rmin ,r2=$r ,r3=$r ,r4=$rmax >> dihedral$r .datecho rk2=200,rk3=200, >> dihedral$r .datecho / >> dihedral$r .datrnext=$(echo "$r + $delta " | bc) sander -O -i md$r .in -o md$r .out -p system.parm7 -c ini$r .rst7 -r md$r .rst7 -ref ini$r .rst7 -x md$n .crd -inf md$r .info cp md$r .rst7 ini$rnext .rst7 ((n++)) done
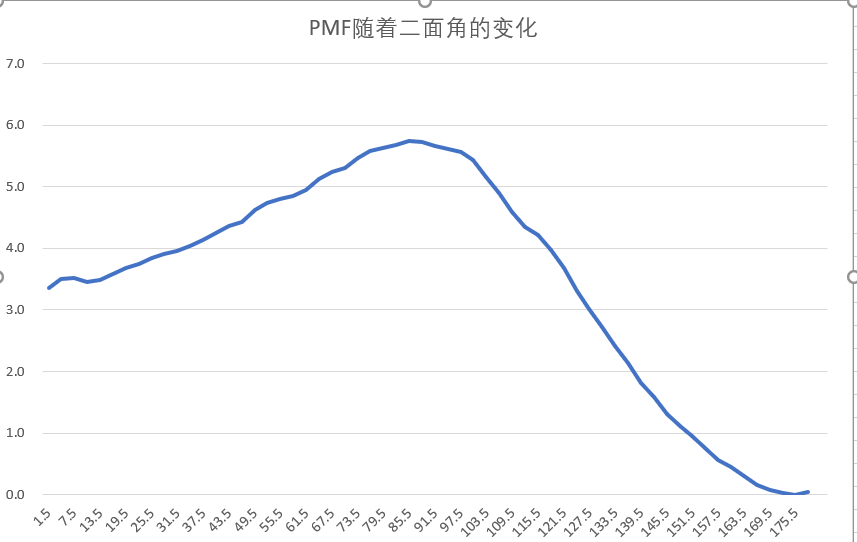
使用WHAM生成PMF
WHAM软件可以去WHAM – Grossfield Lab 下载,安装很简单。meta.dat。此文件有三列,由二面角文件名,二面角值和力常数组成。
Please remember that Amber uses k (x − x0 )2 with k in kcal/mol/rad2 0 )2 with k in kcal/mol/deg2 therefore we need to multiply our force constant by 2(π/180)2 .
可以使用脚本生成neta.dat文件也可以手动完成。
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 use Cwd;$wd=cwd; print "Preparing meta file \n" ;$name="meta.dat" ; $incr=-3 ; $force=0 .12184 ; &prepare_input(); exit (0 );sub prepare_input () $dihed=180 ; while ($dihed>=0 ) { print "Processing dihedral: $dihed \n" &write_meta(); $dihed +=$incr; } } sub write_meta open METAFILE,">>" ,"$name" ; print METAFILE<<EOF; dihedral$dihed.out $dihed.0 $force EOF close MDINFILE; }
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 dihedral180 .out 180.0 0 .12184 dihedral177.out 177.0 0 .12184 dihedral174.out 174.0 0 .12184 dihedral171.out 171.0 0 .12184 dihedral168.out 168.0 0 .12184 dihedral165.out 165.0 0 .12184 dihedral162.out 162.0 0 .12184 dihedral159.out 159.0 0 .12184 dihedral156.out 156.0 0 .12184 dihedral153.out 153.0 0 .12184 dihedral150 .out 150.0 0 .12184 dihedral147.out 147.0 0 .12184 dihedral144.out 144.0 0 .12184 dihedral141.out 141.0 0 .12184 dihedral138.out 138.0 0 .12184 dihedral135.out 135.0 0 .12184 dihedral132.out 132.0 0 .12184 dihedral129.out 129.0 0 .12184 dihedral126.out 126.0 0 .12184 dihedral123.out 123.0 0 .12184 dihedral120 .out 120.0 0 .12184 dihedral117.out 117.0 0 .12184 dihedral114.out 114.0 0 .12184 dihedral111.out 111.0 0 .12184 dihedral108.out 108.0 0 .12184 dihedral105.out 105.0 0 .12184 dihedral102.out 102.0 0 .12184 dihedral99.out 99.0 0 .12184 dihedral96.out 96.0 0 .12184 dihedral93.out 93.0 0 .12184 dihedral90 .out 90.0 0 .12184 dihedral87.out 87.0 0 .12184 dihedral84.out 84.0 0 .12184 dihedral81.out 81.0 0 .12184 dihedral78.out 78.0 0 .12184 dihedral75.out 75.0 0 .12184 dihedral72.out 72.0 0 .12184 dihedral69.out 69.0 0 .12184 dihedral66.out 66.0 0 .12184 dihedral63.out 63.0 0 .12184 dihedral60 .out 60.0 0 .12184 dihedral57.out 57.0 0 .12184 dihedral54.out 54.0 0 .12184 dihedral51.out 51.0 0 .12184 dihedral48.out 48.0 0 .12184 dihedral45.out 45.0 0 .12184 dihedral42.out 42.0 0 .12184 dihedral39.out 39.0 0 .12184 dihedral36.out 36.0 0 .12184 dihedral33.out 33.0 0 .12184 dihedral30 .out 30.0 0 .12184 dihedral27.out 27.0 0 .12184 dihedral24.out 24.0 0 .12184 dihedral21.out 21.0 0 .12184 dihedral18.out 18.0 0 .12184 dihedral15.out 15.0 0 .12184 dihedral12.out 12.0 0 .12184 dihedral9.out 9.0 0 .12184 dihedral6.out 6.0 0 .12184 dihedral3.out 3.0 0 .12184 dihedral0 .out 0 .0 0 .12184
运行wham软件
1 ./wham P 0 180 60 0.01 300 0 meta.dat result.dat
P:反应坐标是周期性的
1 2 3 4 cat result.dat | awk "{print $1 ,$2 }" > pmf.dat xmgrace pmf.dat
后话
因为自身过于懒惰,本来想把该教程写的更加详实以及直白,但是偷懒了,最后也就这样了。在此根据该教程的使用软件情况给自己挖几个博客的坑.什么时候能够填上?有谁会知道呢?
perl/bash语言使用教程wham、xmgrace/qtgace安装教程—>极致简单TCL语言开发VMD插件